ゲノミクスラーニングセンター
生命科学分野の1つ「ゲノミクス(Genomics)」に関連する新たな研究手法を解説します。最新の技術によって、複雑な生物システムを解き明かすために、細胞懸濁液や組織サンプル中の1細胞レベルでの知見を得ることができます。

ゲノミクス分野に関連する最新アプリケーション
ゲノムおよび遺伝子を研究対象とする「ゲノミクス」分野において、RNAを研究対象とする「トランスクリプトミクス(Transcriptomics)」やタンパク質を研究対象とする「プロテオミクス(Proteomics)」等を含む、複数の階層にわたる様々なデータを組み合わせて実施される「マルチオミクス(Multiomics:複数のオミクス)」解析を用いたアプローチの重要性は一段と増しています。例えば、最新の研究手法である「シングルセルRNA-seq解析(scRNA-seq:Single-cell RNA sequencing)」や「空間トランスクリプトーム解析(ST:Spatial transcriptomics)」等を併用するマルチオミクス的アプローチによって、特定の細胞種別(細胞のアイデンティティ)とその機能に関する洞察を得ることができます。これらのデータを活用すると、多種多様な細胞群で構成される複雑なシステムの全体像をより俯瞰的に捉えることが可能になります。さらに、バーコード配列が付加されたオリゴヌクレオチド標識抗体を使用して、細胞表面タンパク質の定量的・定性的発現情報を取得する「CITE-seq/REAP-seq」と呼ばれる手法の開発をはじめ、各種技術の進歩によりシングルセルRNA-seq(scRNA-seq)解析と並行して、様々なタンパク質の同時測定(「プロテオーム解析」)が可能になりました。シングルセルRNA-seq(scRNA-seq)や空間トランスクリプトーム解析と同時にタンパク質の発現評価も実施することは、以下の利点を提供します。
細胞特性解析の高度化
タンパク質は、細胞内で様々な生物学的プロセスを実行する機能性分子です。mRNAの発現に加えタンパク質の発現を評価することで、細胞の状態/表現型、機能をより包括的に理解することができます。

RNAとタンパク質レベルの比較/検証の実施
シングルセルRNA-seq(scRNA-seq)はmRNAレベルでの遺伝子発現情報を提供する一方、実際に翻訳されたタンパク質の存在量を直接的に示すわけではありません。そのため、scRNA-seq解析データが必ずしもタンパク質レベルを反映しているとは限りません。したがってタンパク質の発現を同時に測定することは、RNA発現レベルとタンパク質発現レベルの相関性や一致度を明らかにし、細胞の状態/表現型をより正確に評価することにつながります。
バイオマーカー探索プロセスの向上/容易化
シングルセルRNA-seq(scRNA-seq)とタンパク質の測定を組み合わせることは、様々な細胞の種類、病態、治療反応に関連するタンパク質マーカーの探索・同定プロセスを容易化します。新規タンパク質マーカーの同定や既存タンパク質マーカーのモニタリングは、疾病の診断、予後予測、治療方針の示唆に寄与します。
シングルセルRNA-seq(scRNA-seq)解析とは?
「シングルセルRNA-seq(scRNA-seq)」または「シングルセルトランスクリプトーム解析」は、単一細胞レベル(1細胞ごと)の解像度で遺伝子発現を網羅的に解析できる非常に優れた最新の手法です。シングルセルRNA-seq(scRNA-seq)では、1回の実験で最大数百万個の細胞のトランスクリプトームプロファイルを得ることができます。シングルセルRNA-seq(scRNA-seq)は、細胞種別(細胞のアイデンティティ)、細胞の挙動、疾病のメカニズム、各種バイオマーカーに関する貴重な手がかりを提供するため、幅広い分野の研究者にとって有力なツールとなっています。シングルセルRNA-seq(scRNA-seq)は、主に以下の利点を提供します。
細胞「不均一性」解明への貢献
scRNA-seqは、多種多様な細胞種が含まれ、複雑な構成をなす細胞混合物中における細胞の種類、亜集団、稀な細胞集団の同定および特性解析を可能にします。
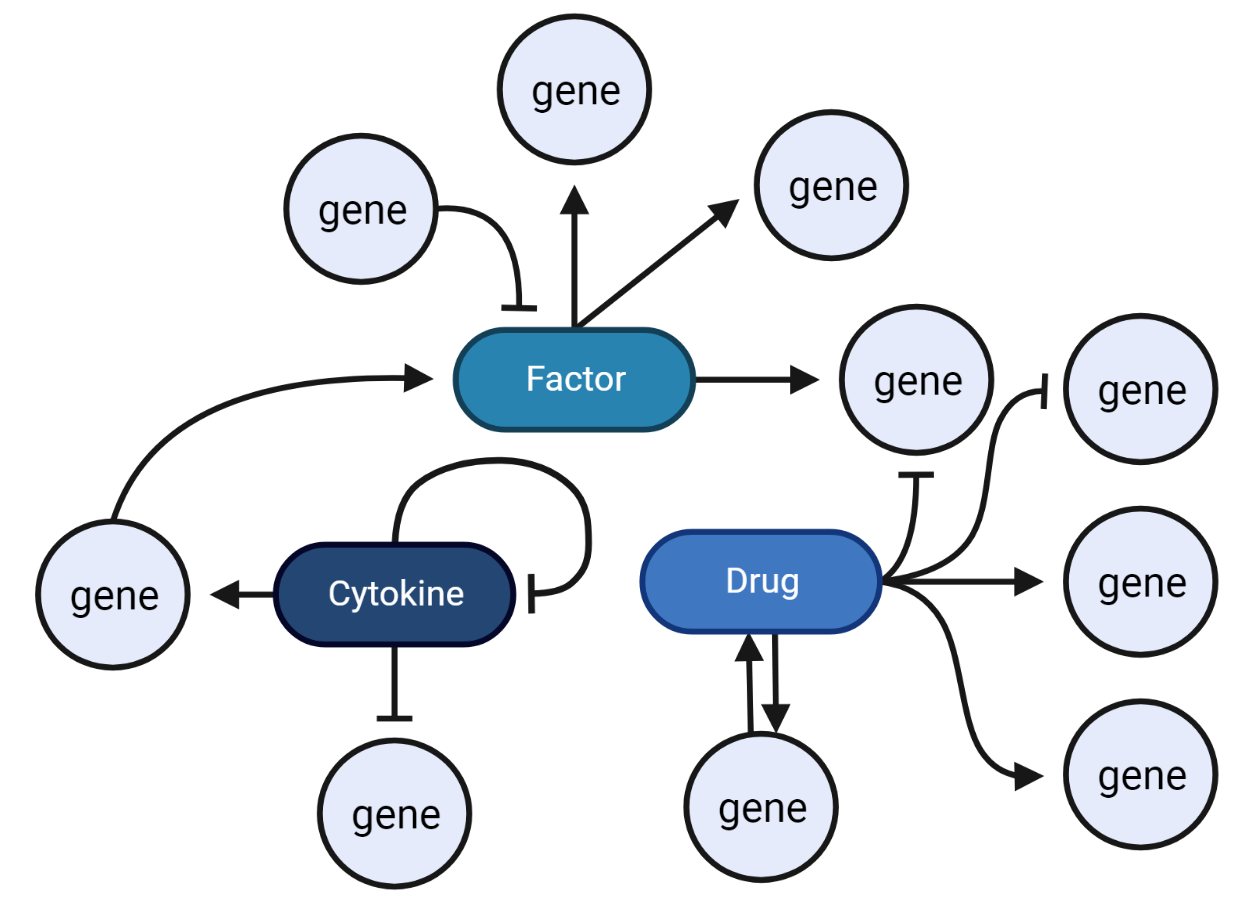
転写ダイナミクス調査の実施
遺伝子制御ネットワーク、細胞運命の決定機構、細胞分化機序の解明、外部刺激に対する細胞応答の理解につながります。
バイオマーカー探索/プレシジョンメディシン(精密医療)開発への寄与
多種多様な各細胞集団に対するマーカーの探察や同定、疾患状態に特異的な新規バイオマーカーの開発に寄与します。このことは、患者個人ごとに最適化された治療を選択できる精密医療の将来的実現につながる可能性があります。
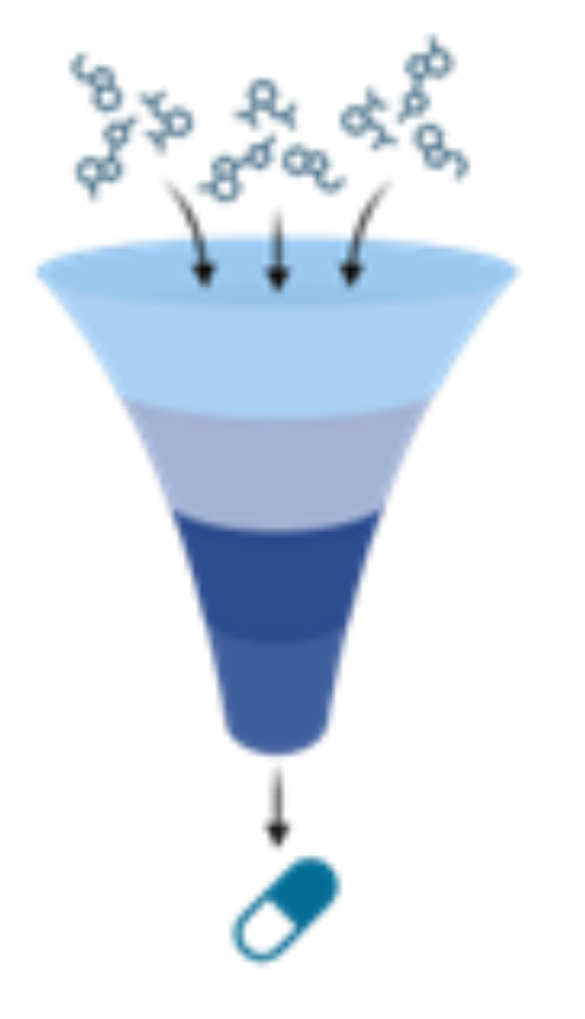
創薬プロセス・医薬品開発プロセスでの薬効評価における応用可能性
薬物応答に関連した高深度かつ高精度な解析を実現し、創薬・医薬品シーズの作用機序の解明、治療薬候補品の有効性および安全性の評価に応用できる可能性があります。
シングルセルRNA-seq(scRNA-seq)解析のワークフロー
- 細胞懸濁液を調製します。
- 適切な手法により、細胞懸濁液に含まれる各細胞をシングルセル(単一細胞)に分画化します。

- 細胞溶解処理または細胞透過処理を実施します。本処理により、単一細胞内のmRNAおよびその他成分は、下流の分子生物学的手法による解析に利用できるようになります。
- 逆転写反応により、mRNAからcDNAを生成します。逆転写反応の過程では、由来となる単一細胞を識別するためのIDとして、固有のバーコード(「セルバーコード」)を付加します。すなわち、それぞれの単一細胞から調製される逆転写産物(cDNA)には、それぞれの単一細胞画分ごとに異なる個別の「セルバーコード」が付与されることになります。

- セルバーコードが付与されたcDNAをプールします。その後、適切な方法によりシーケンシング用の二本鎖DNAライブラリーを調製します。

- 次世代シーケンサーを利用した次世代シーケンシング解析(NGS)により、二本鎖DNAライブラリーに含まれる単一細胞由来の転写産物およびセルバーコードの塩基配列を読み取ります。

- 次世代シーケンシング解析により得られたデータ(NGSデータ)は、単一細胞ごとの遺伝子発現状況(転写産物/mRNA発現レベル)を反映します。得られたNGSデータを使用し、個々の細胞の表現型/機能の解析を実施します。
シングルセル(単一細胞)分画化/scRNA-seq解析方法
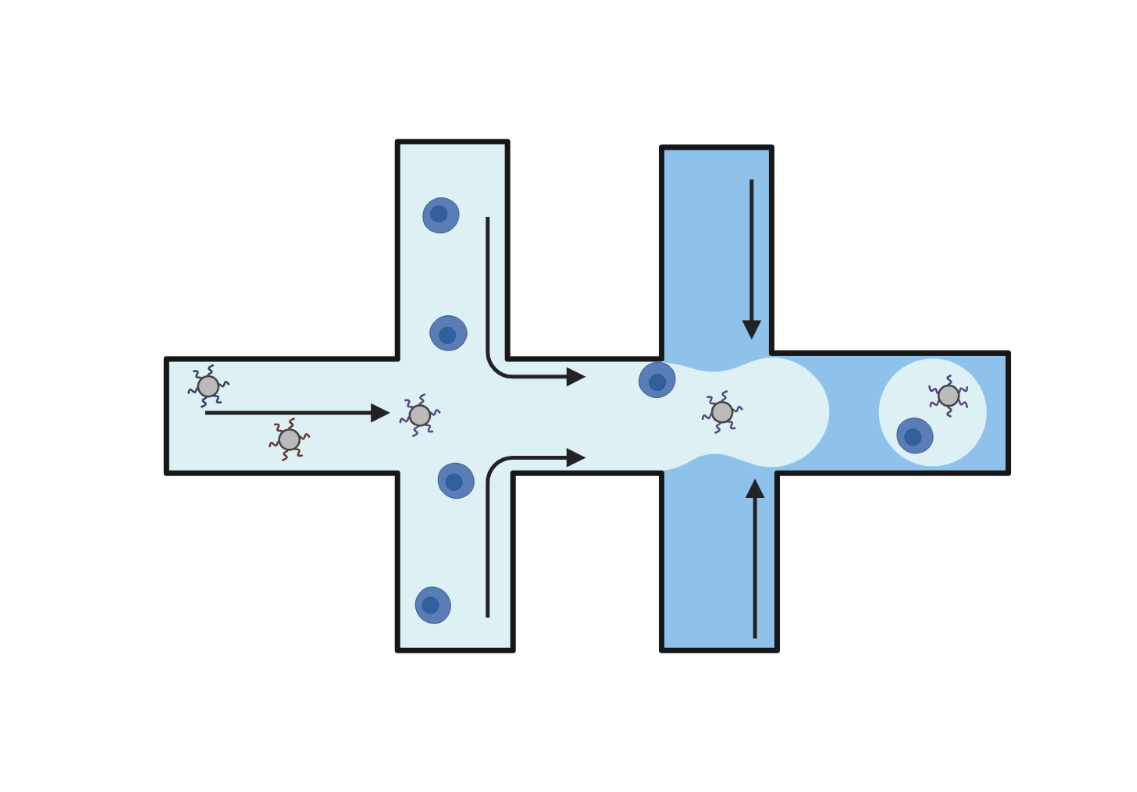
ドロップレット(Droplet)法
ドロップレット法では、マイクロ流体力学/マイクロ流路技術を利用して予めバーコード配列が付与されているビーズ(ゲルビーズ)と単一細胞(シングルセル)を合流させた後、油中に分散する水性の液滴(ドロップレット/エマルジョン)を形成させると共に、1つの液滴内に個々のビーズと単一細胞を封入します。続いて、各液滴内で細胞を溶解させ、逆転写反応を実施することでバーコード化cDNAを調製します。バーコード化cDNAをプールして次世代シーケンシング解析(NGS)用ライブラリーを調製後、次世代シーケンサーを用いて塩基配列を読み取ります。ドロップレット法では、1回の実験で最大100万個の細胞を分析対象にできるため、ハイスループットな解析が可能となります。
マイクロウェル(Microwell)法
マイクロウェルを用いる方法では、単一細胞(シングルセル)単離用の微細構造(マイロウェルアレイ)を有する特殊なカートリッジ型デバイスを使用します。それぞれの細胞は、mRNAキャプチャービーズと共に別々のマイクロウェルに隔離されます。各ウェルの中で、細胞溶解、逆転写、バーコード付与の処理は実施されます。その後、プール化したバーコード化cDNAライブラリーを用いて、次世代シーケンサーにより塩基配列を決定します。
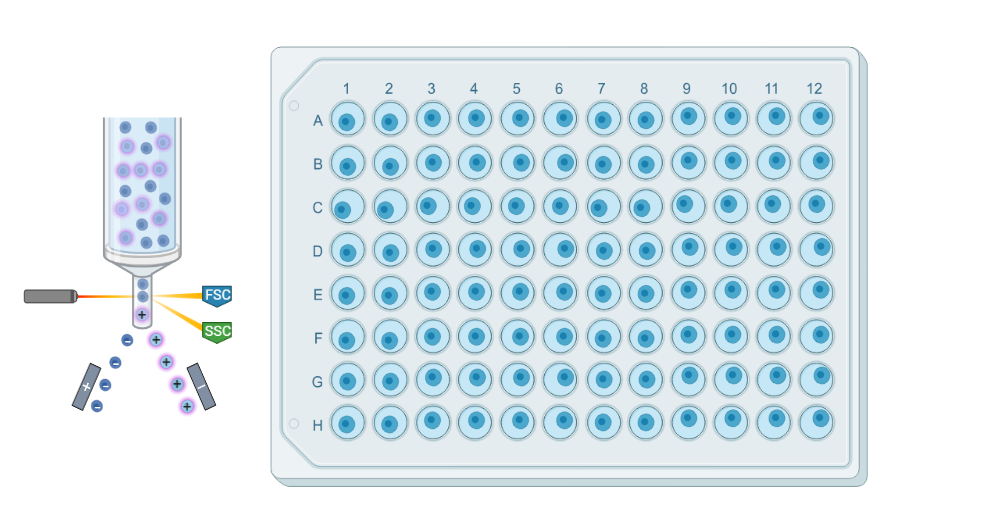
プレートベース(Plate-based)法
プレートを用いたscRNA-seq法の場合、セルソーティングによって細胞を分離後、従来型のプレートフォーマットで個々の細胞を調製します。通常、96ウェルプレートまたは384ウェルプレートに単一細胞が区画化され、逆転写およびライブラリー調製は各ウェルの中で実施されます。
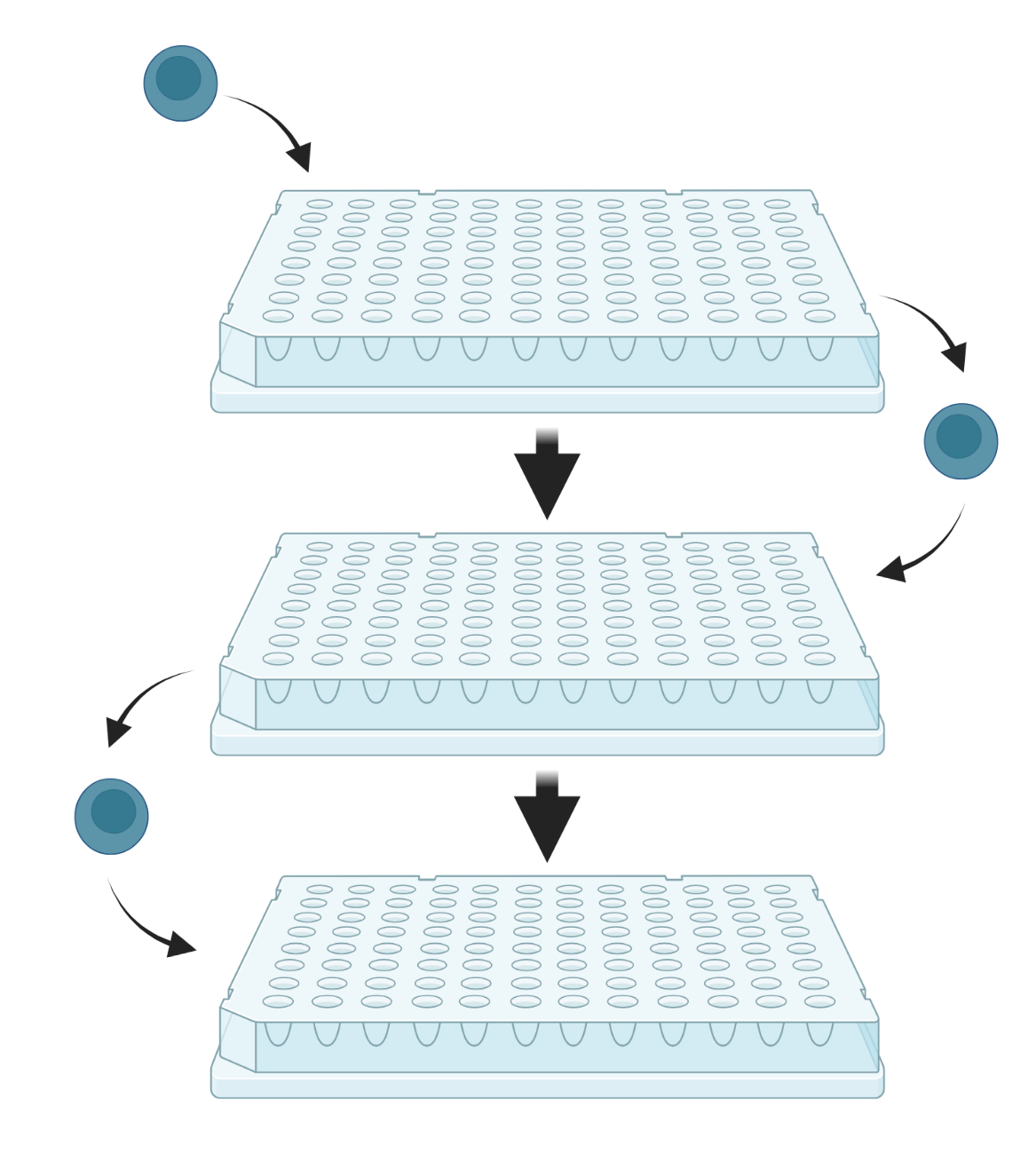
スプリットプール(Split-pool)法
スプリットプール法では、主に固定細胞を対象として、複数のマルチウェルプレートを介して細胞の「スプリット(分配)」と「プール(混合)」を繰り返し行う、多段階的なセルバーコードの付与を特徴とします。本手法は「split and pool」「コンビナトリアルバーコーディング/組み合わせバーコーディング(combinatorial barcoding)」「SPLiT-seq(Split Pool Ligation-based Transcriptome sequencing)」等とも呼ばれる、シングルセルトランスクリプトーム解析手法の1つです。各プレートの各ウェルにはウェル固有の異なる配列のオリゴヌクレオチドバーコードが含まれており、細胞がウェルからウェルへと連続的に移動するにつれてユニークなバーコード化が実行されます。数ラウンドの「スプリット」と「プール」を繰り返して得られたバーコード化cDNAを使用して、次世代―シーケンサーにより塩基配列を決定します。
空間トランスクリプトーム解析とは?
空間トランスクリプトーム/空間トランスクリプトミクス解析(ST:Spatial Transcriptomics)とは、組織構造/形態や細胞位置等の空間的情報(空間的コンテキスト)を保持した状態で、転写産物(トランスクリプトーム)の解析結果を重ね合わせることにより、空間的な遺伝子発現状態の可視化および定量化を実施するための一連の方法論を指します。シングルセルRNA-seq(scRNAseq)解析等のトランスクリプトーム解析技術の場合、単一細胞レベルでの豊富な遺伝子発現情報を取得できますが、対象遺伝子が組織のどの部位で発現しているかという空間的情報は失われてしまっています。一方、空間トランスクリプトーム解析の場合、次に述べる主要な2通りの手法によって、空間的情報の取得に関わる制約に対処することが可能となります。(1)1つ目は、オリゴヌクレオチドが連続的に配置された特殊なスライドを用いて、空間バーコード/空間キャプチャー技術により、mRNAを捕捉すると共に物理的・空間的座標(「空間バーコード配列」)を付与した後、次世代シーケンシング解析を実施する方法です。(2)2つ目として、smFISHと呼ばれる技術をベースにしたMERFISHやseqFISH+等のように、標的とするmRNA分子に対してそれぞれユニークな「読み取り(リードアウト)配列」が含まれる遺伝子特異的プローブを直接的にハイブリダイズした後、読み取り配列に対する各蛍光プローブのハイブリダイズと脱ハイブリダイズを連続的に実施することで、in situで各mRNAの発現分布を可視化する方法が挙げられます。空間トランスクリプトーム解析は、以下に示すいくつかの理由により徐々に普及しつつあります。
空間的情報(空間的コンテキスト)を取得可能
空間的情報を維持した状態で遺伝子発現の包括的全体像を得ることができます。組織内の細胞は、周囲の細胞との接触や環境因子の影響を受ける複雑な環境下に存在するため、空間的情報の取得は極めて重要です。
複雑な組織を細胞レベルで理解可能
空間トランスクリプトーム解析では、細胞レベルの遺伝子発現情報を入手できるため、脳や腫瘍といった複雑な組織の解析に有用です。
疾患研究に寄与する可能性
空間トランスクリプトーム解析は、がん等の疾患研究で特に有用な手法と考えられています。がん細胞と周辺組織中の細胞間での相互作用や、腫瘍微小環境の解明につながる可能性があります。
創薬プロセスでの活用への期待
組織内/腫瘍内の異なる領域が特定の薬物治療にどのように応答するかを同定する一助となり、創薬に寄与する可能性があります。
シングルセルレベルの解像度でデータ取得
いくつかの空間トランスクリプトーム解析手法は、技術革新により、シングルセルレベルまたは細胞内レベルでの遺伝子発現解析を達成しています。高解像度な遺伝子発現解析は、特定の空間的環境下に存在する個々の細胞ごとの詳細な遺伝子発現情報の取得を可能にします。
代表的な空間トランスクリプトーム解析手法
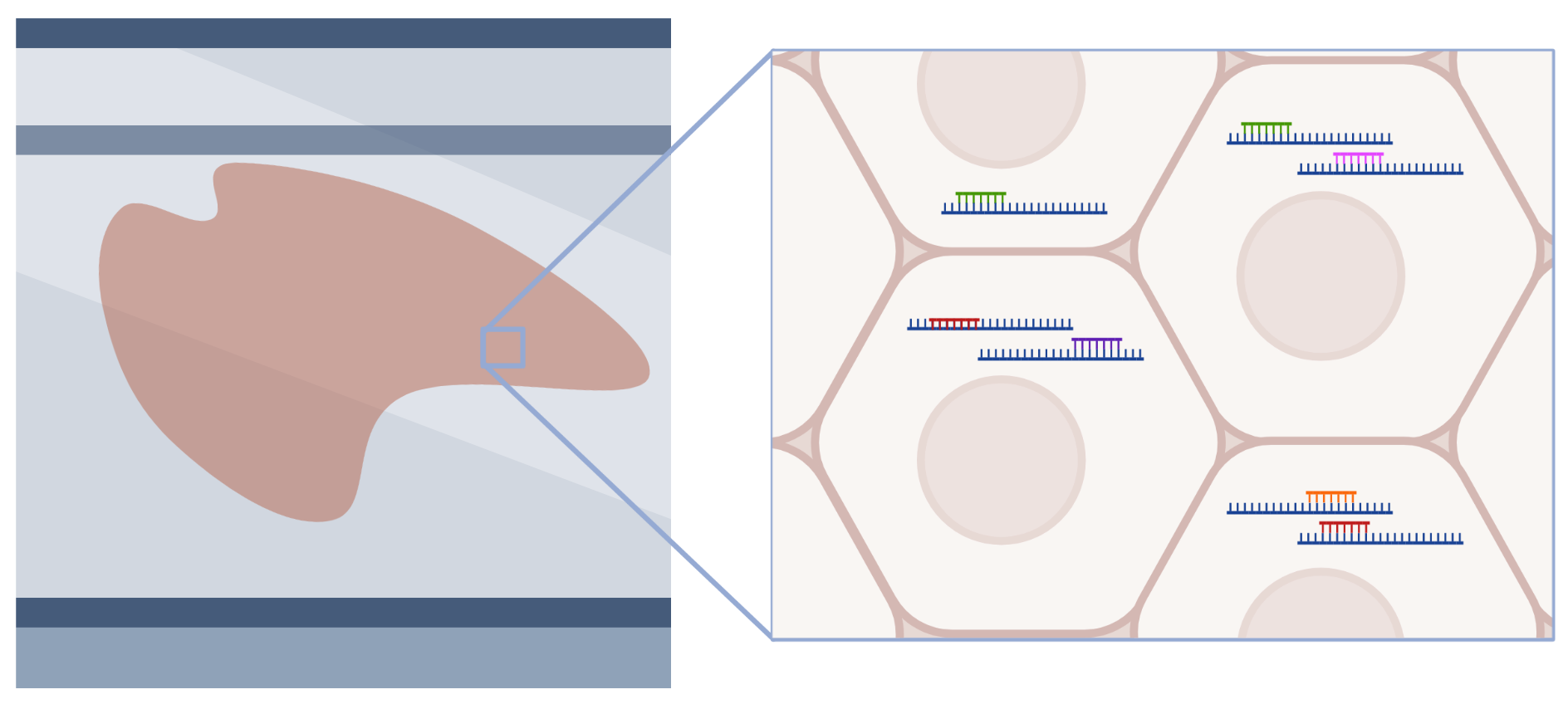
1. 空間バーコード/空間キャプチャー技術を利用したRNAシーケンシング(RNA-seq)
本手法ではDNAオリゴヌクレオチドで被覆された特殊なスライドガラス上に組織切片を配置します。スライドガラス上の各DNAオリゴヌクレオチドには固有の空間バーコード配列が含まれており、組織切片をスライドガラスに配置すると、組織中のmRNAは固有の空間バーコード配列を有するDNAオリゴヌクレオチドによって捕捉されます。その後、mRNAを逆転写してスライドから取り出した後、次世代シーケンシング解析用ライブラリーを調製します。次世代シーケンサーによる塩基配列決定を行い、取得された空間バーコード情報を利用することで、各mRNA分子が存在した組織内の位置まで遡って、発現遺伝子の空間的情報を追跡することができます。
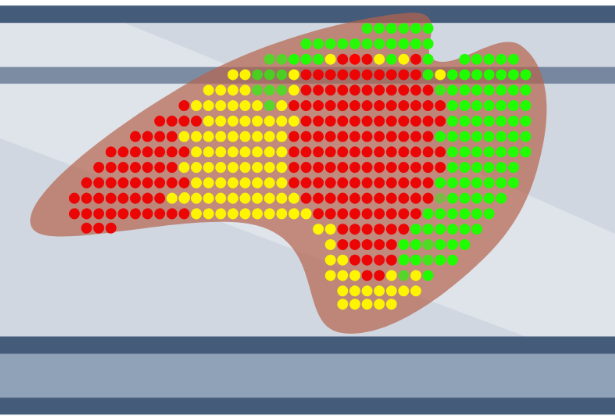
2. 蛍光in situ hybridization(FISH)
スライドガラスに組織切片をマウントします。ターゲットmRNAに特異的な各プローブのプールに暴露し、各プローブを組織切片中のmRNAにハイブリダイズします。その後、それぞれのプローブは「コンビナトリアルラベリング/組み合わせラベリング(combinatorial labeling)」的アプローチに基づいて同定されます。つまり、各プローブには固有の「読み取り(リードアウト)配列」が含まれており、読み取り配列に対する各蛍光プローブのハイブリダイズ・イメージング・脱ハイブリダイズの連続工程を複数回繰り返すことによって、特定のmRNAにハイブリダイズしたプローブを同定します。このアプローチにより、数百から数千種類のmRNA分子をシングルセルレベルまたは細胞内レベルの解像度でプロファイリングすることが可能となります。
1. 空間バーコード/空間キャプチャー技術を利用したRNA-seqのワークフロー
- FFPE(ホルマリン固定パラフィン包埋)組織切片または新鮮凍結組織切片を専用スライドに移します。専用スライド上には空間バーコード配列を含有するDNAオリゴヌクレオチドが配置されており、組織切片中のmRNAを捕捉(キャプチャー)およびバーコード化します。
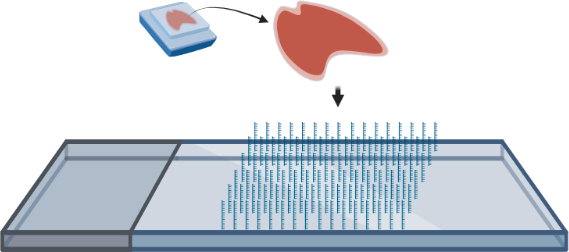
- 組織を透過処理して、mRNAをDNAオリゴヌクレオチドアレイ上に漏出させます。
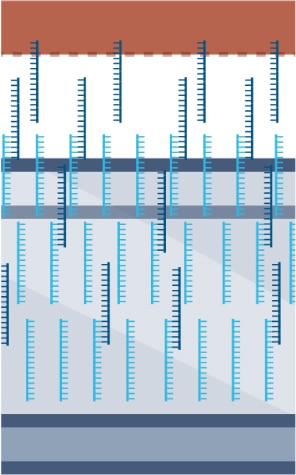
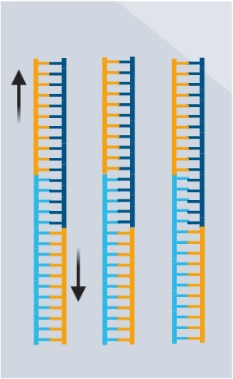
- mRNAを逆転写・伸長させます。
- 得られたcDNAを回収し、次世代シーケンシング用の二本鎖DNAライブラリーを調製します。

- 次世代シーケンサーを用いてライブラリーの塩基配列を読み取ります。

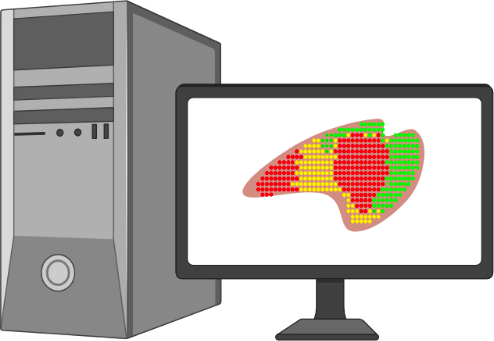
- データ解析を実施し、DNAオリゴヌクレオチドの空間バーコード情報に基づき、各mRNA発現を元のDNAアレイ/組織上の特定位置にマッピングします。
2. 蛍光in situ hybridization(FISH)のワークフロー
- FFPE(ホルマリン固定パラフィン包埋)組織切片または新鮮凍結組織切片を顕微鏡観察用スライドガラス上にマウントし、発現遺伝子に特異的なプローブで染色するための前処理を行います。
- 各転写産物(mRNA)に特異的なプローブで組織を染色します。各プローブには解析に利用される「読み取り(リードアウト)配列」が含まれます。次のステップで示される連続的イメージングは、蛍光標識されたレポータープローブを読み取り配列に結合させることで実施されます。
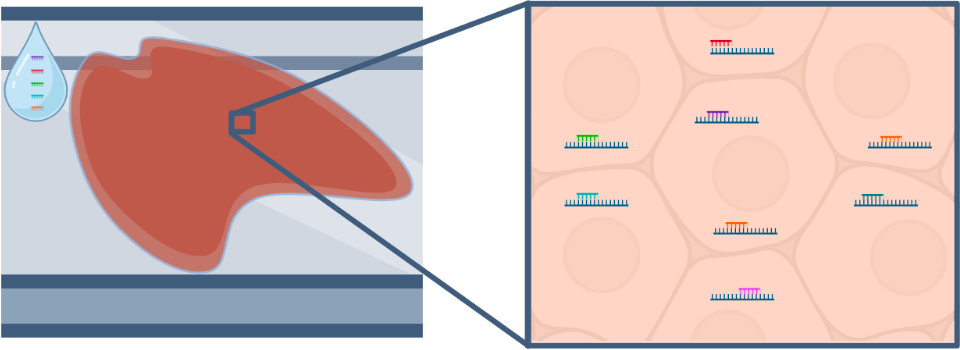
- 顕微鏡を用いて「レポータープローブ結合、イメージング、洗浄」の一連の工程を繰り返すことでmRNAを同定します。つまり、複数ラウンドのイメージング過程で読み取られたプローブ固有の連続的シグナル情報を取得することで、mRNAを識別します。
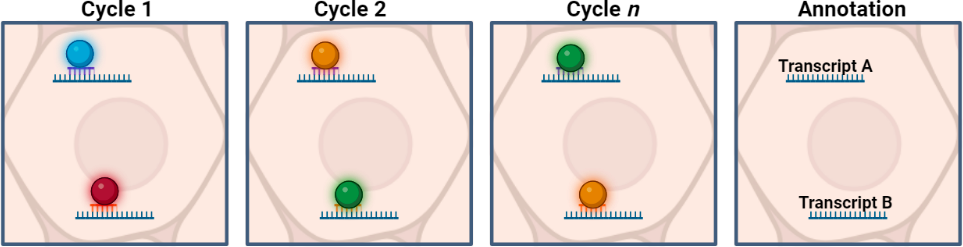
- データ解析によって、細胞のセグメンテーションや細胞の分類が可能になります。